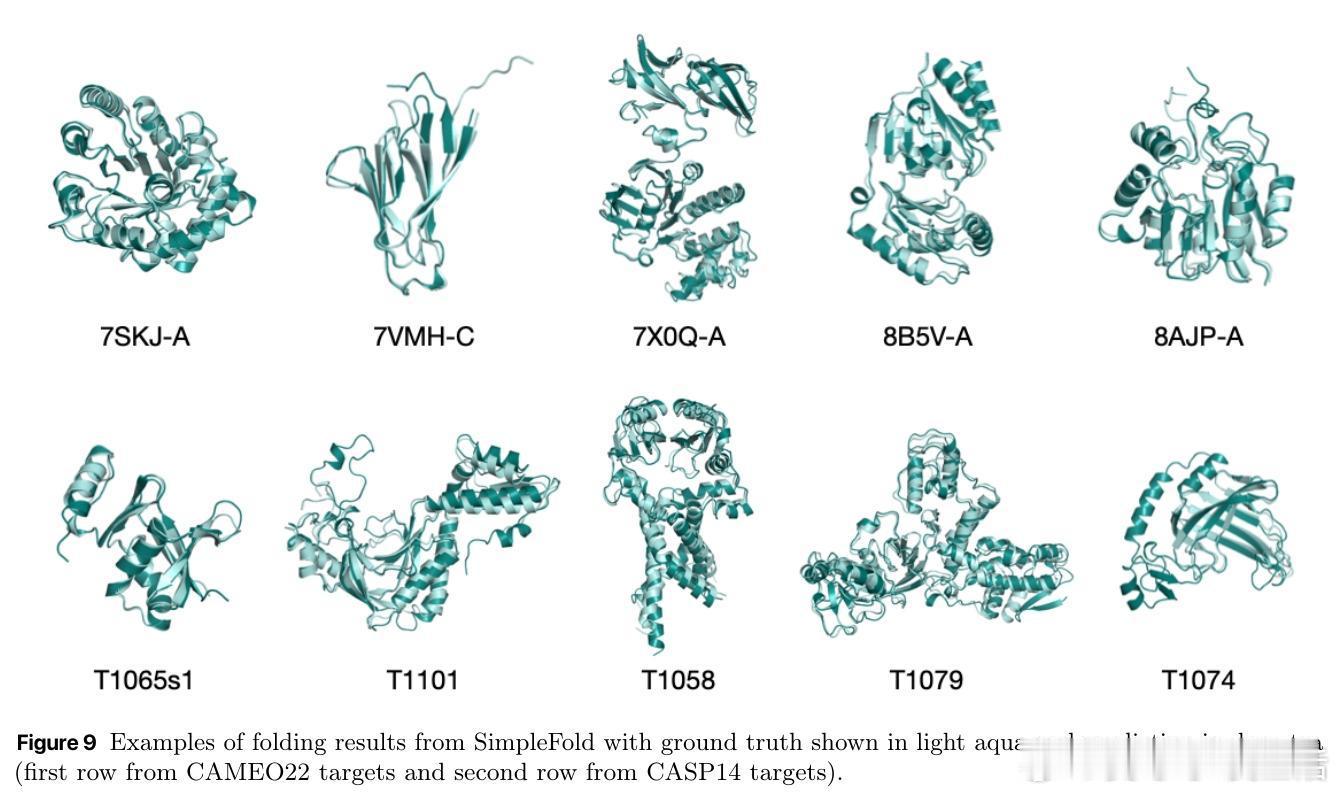
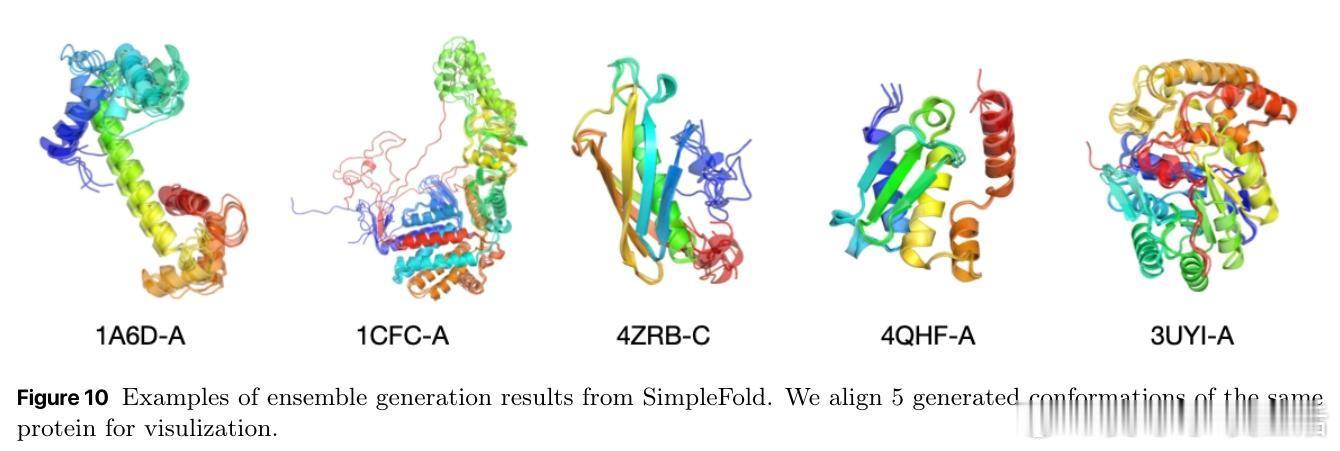
[LG]《SimpleFold: Folding Proteins is Simpler than You Think》Y Wang, J Lu, N Jaitly, J Susskind... [Apple] (2025)
SimpleFold:用通用Transformer简化蛋白质折叠,性能媲美顶尖模型
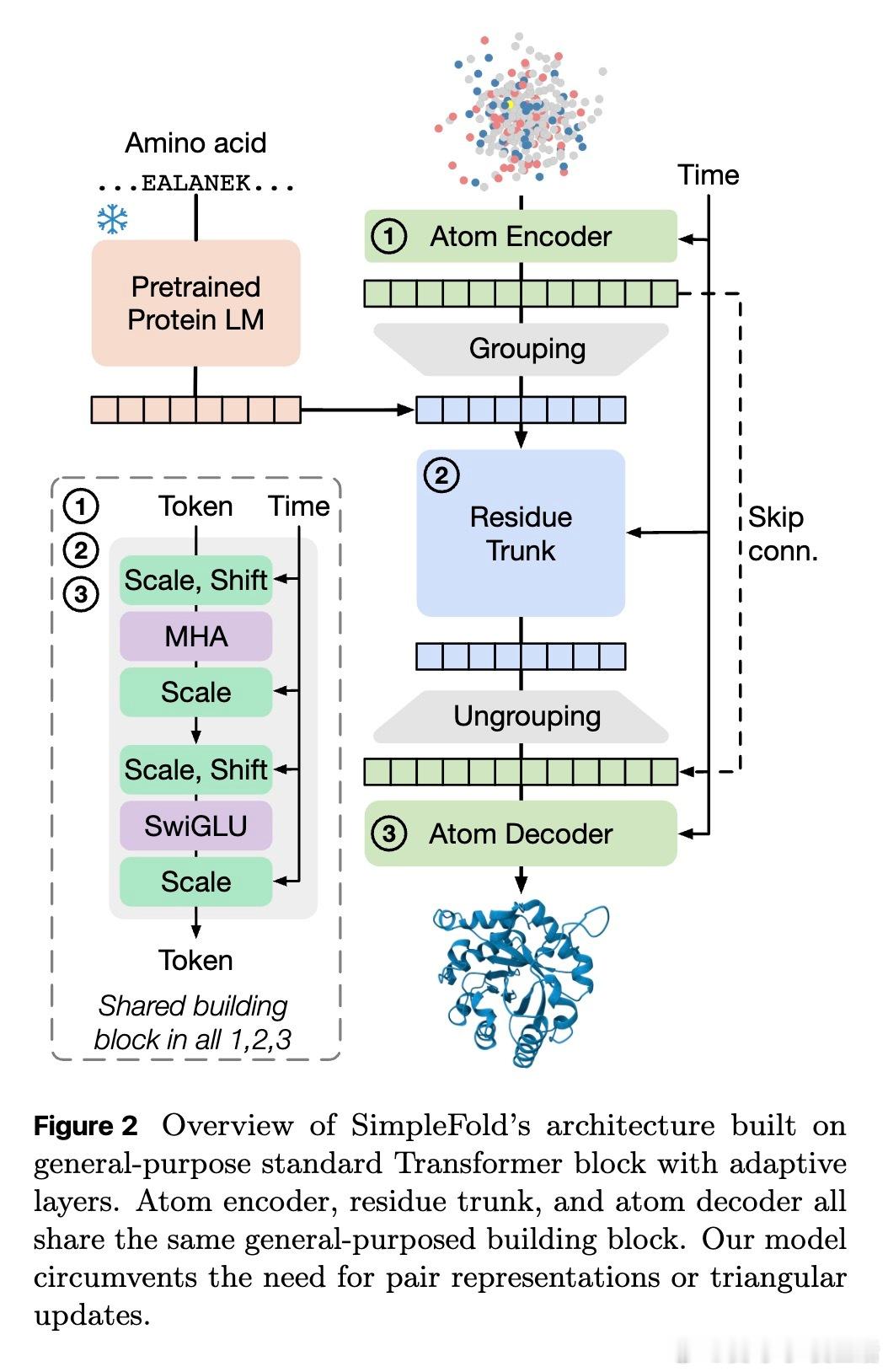
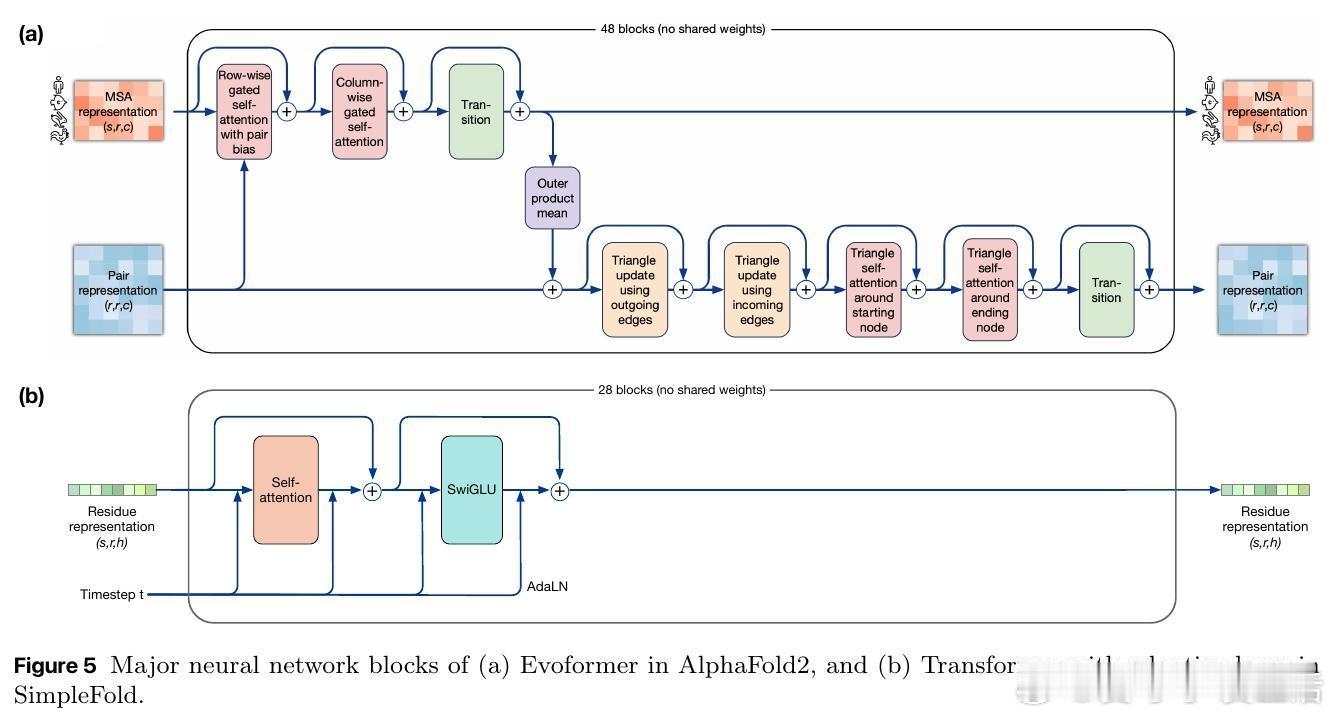
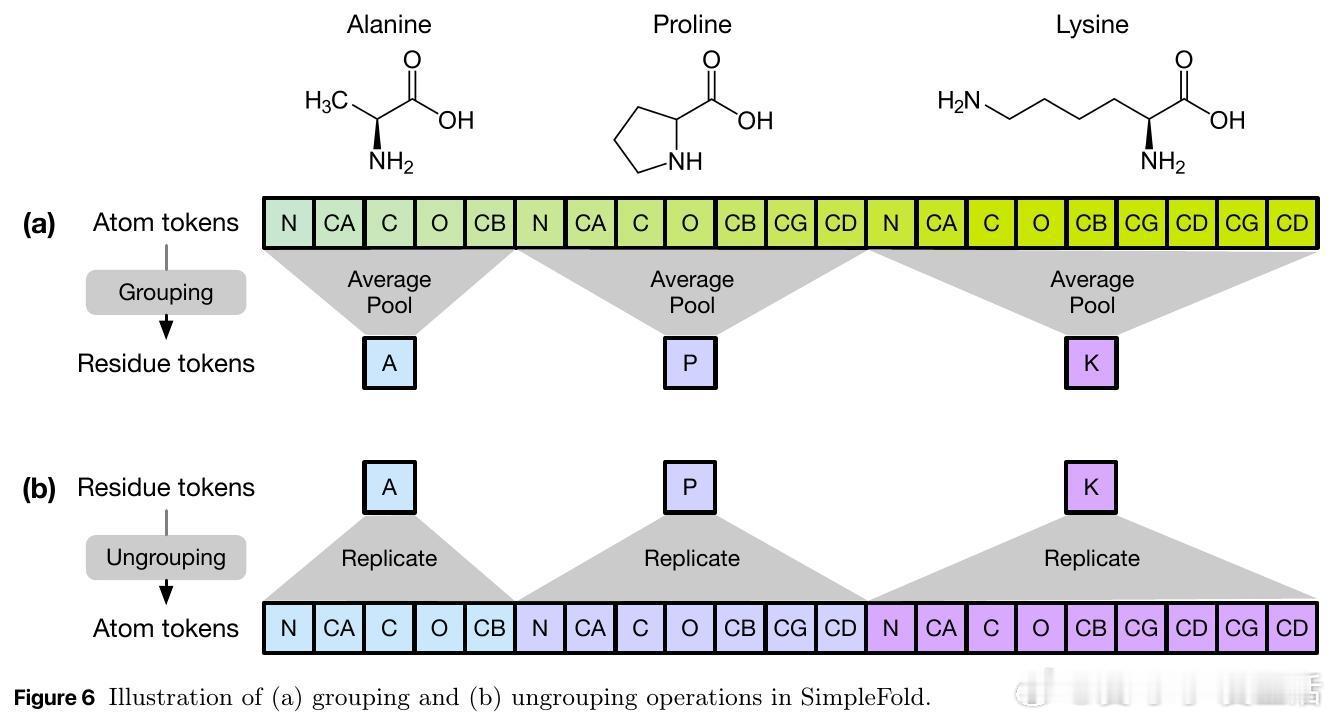
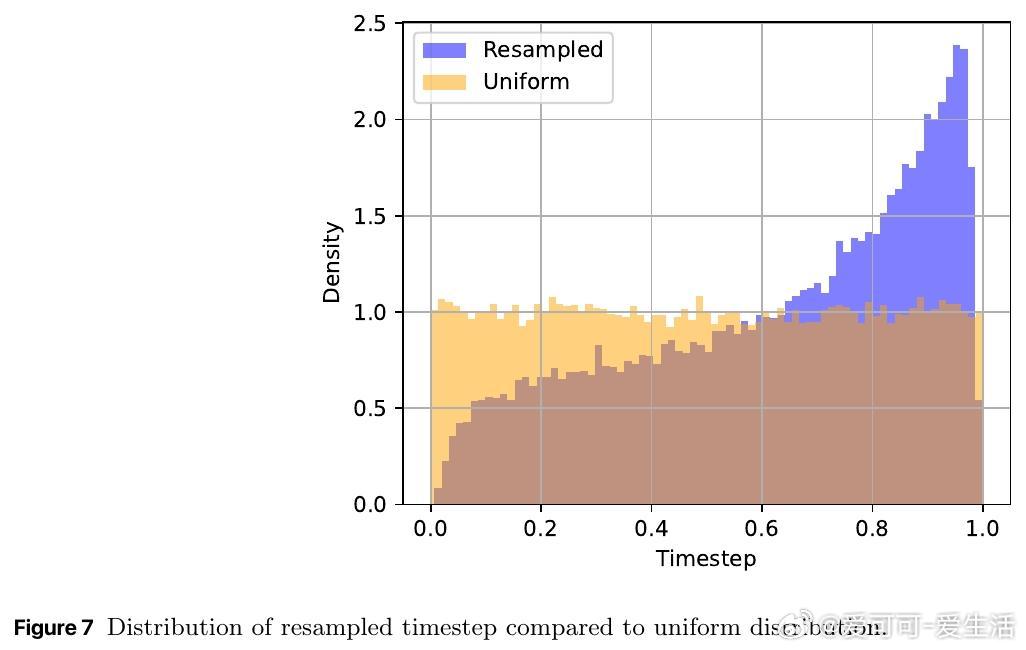
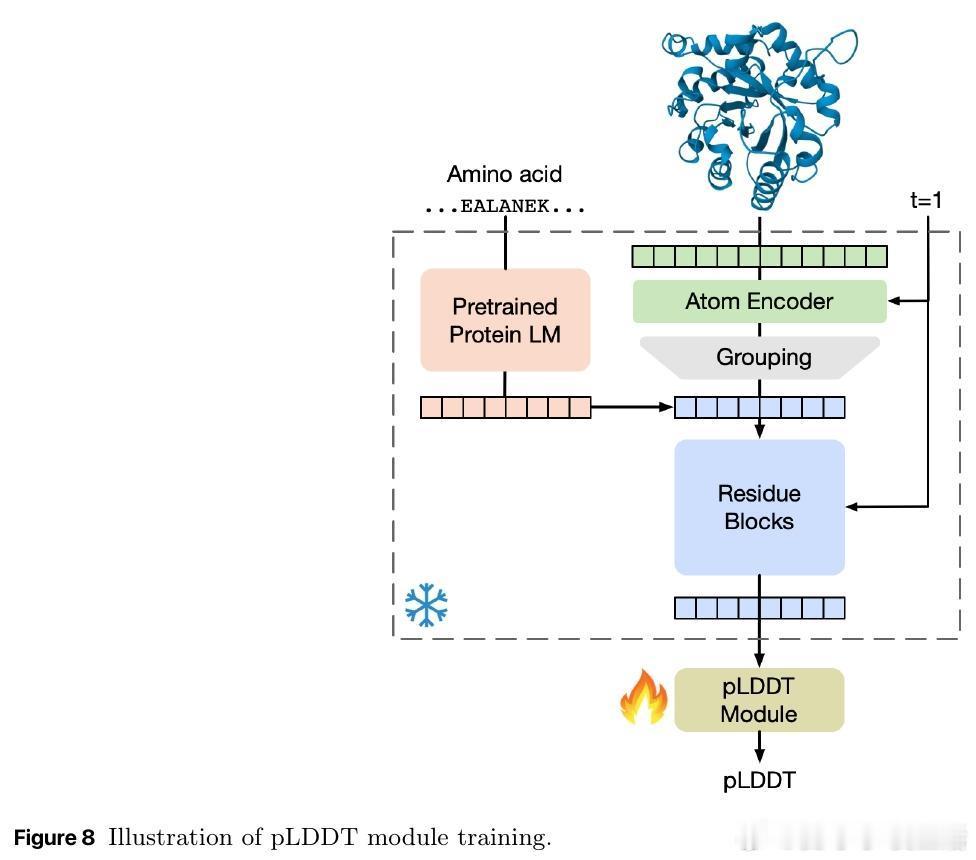
• 创新点:首个基于flow-matching的蛋白质折叠生成模型,摒弃复杂领域特定模块(MSA、多序列比对、三角更新、对偶表示),仅用通用Transformer块+自适应层。
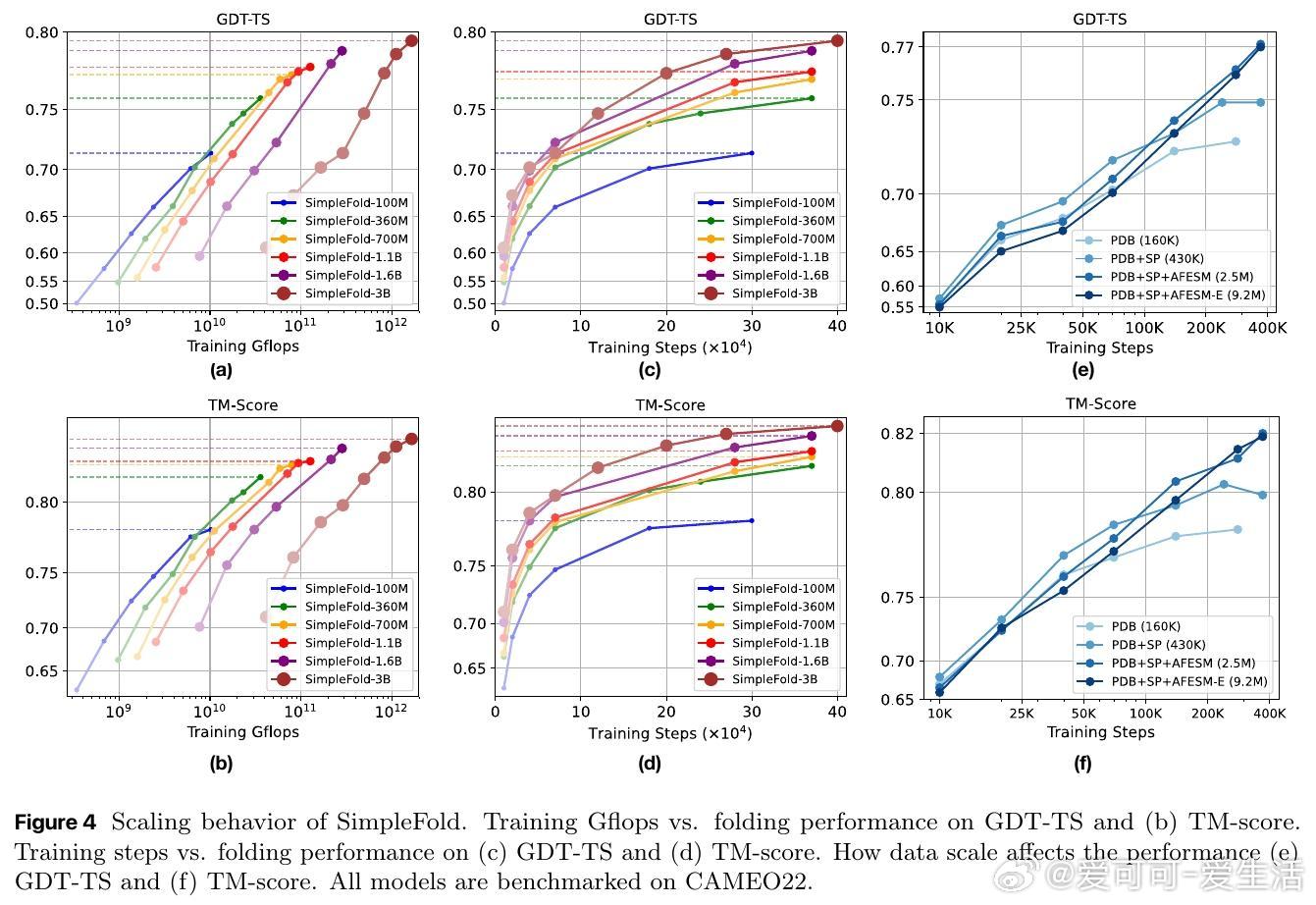
• 规模与数据:模型最大达30亿参数,训练数据涵盖约900万蒸馏结构及实验PDB,充分利用海量蛋白质结构信息。
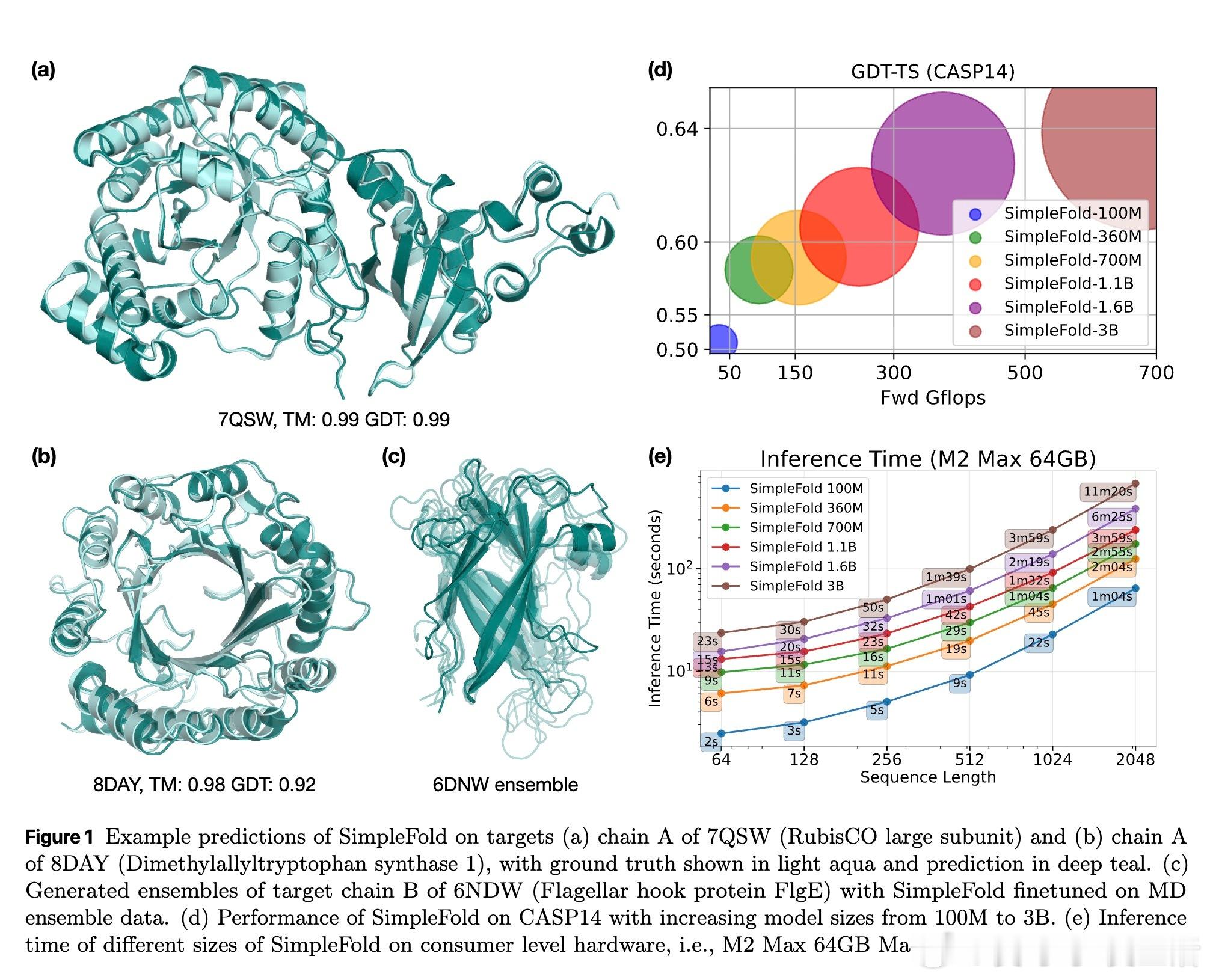
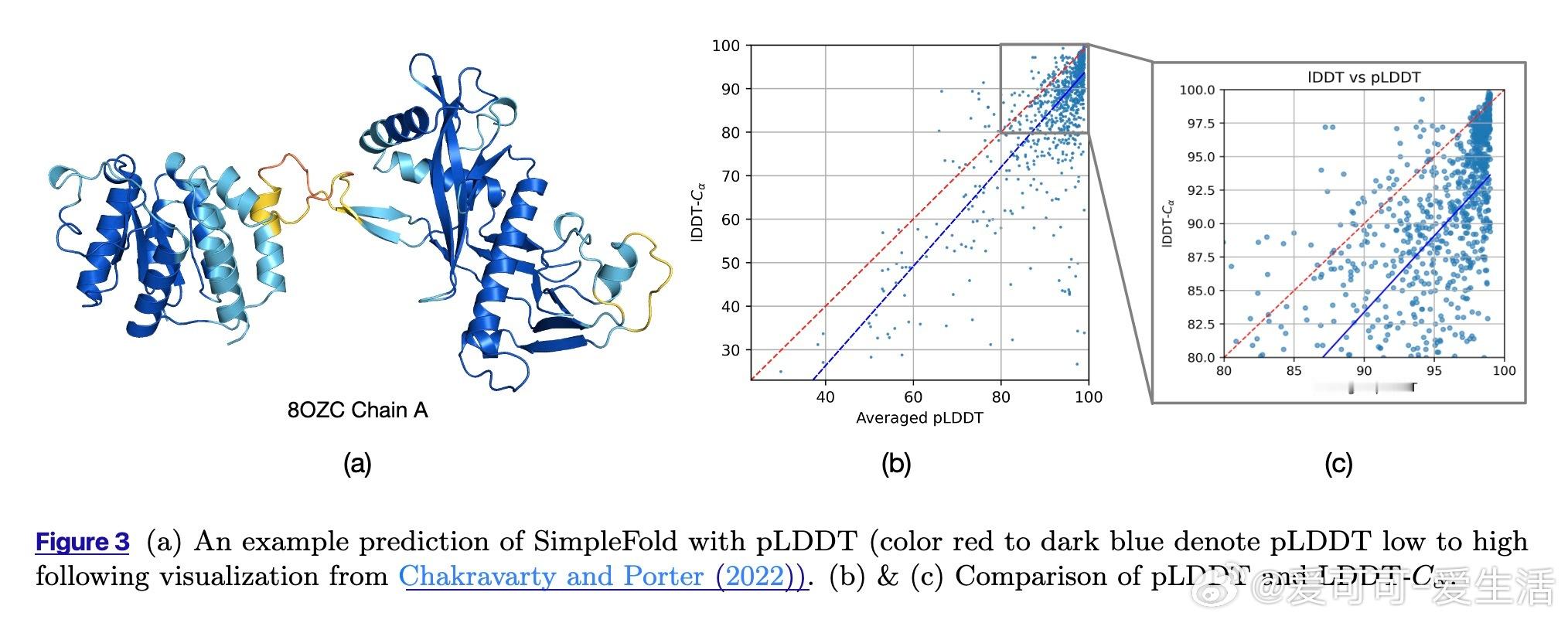
• 性能表现:在CAMEO22和CASP14等权威基准测试中,SimpleFold-3B表现接近AlphaFold2和RoseTTAFold2,尤其在复杂任务CASP14展现更强鲁棒性。
• 生成多构象:作为生成模型,SimpleFold天然支持蛋白质结构多样性建模,优于传统回归模型,具备分子动力学MD模拟数据拟合能力,支持多态结构预测。
• 计算效率:相比AlphaFold2,SimpleFold架构简洁,推理时计算资源需求显著降低,100M参数版本即可在消费级硬件快速部署。
• 设计理念突破:挑战蛋白质折叠领域对复杂启发式架构的依赖,证明规模化训练和通用架构足以捕获蛋白质结构空间的对称性和复杂性。
心得:
1. 简化架构与大规模数据训练相结合,可有效捕获蛋白质折叠的本质规律,减少设计偏见带来的局限。
2. 生成式建模自然支持结构的多样性,拓展了蛋白质结构预测向动态构象群的方向,契合生物分子的实际生理状态。
3. 通用Transformer的成功应用为蛋白质折叠及相关生物分子建模领域开辟了新路径,未来可借力预训练语言模型进一步提升结构表征能力。
代码开源,助力生物信息学和药物设计领域创新发展。
详情🔗 arxiv.org/abs/2509.18480
蛋白质折叠生成模型Transformer生物信息学计算生物学